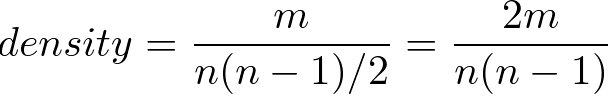[pig][メモ]DataFuにあるPageRankを試してみた
Pig UDFのライブラリDataFuに、PageRankがあるらしい事を知ったので試してみた。
・http://twitter.com/shiumachi/status/253478760119156736
ライブラリのダウンロードと展開
$ wget --no-check-certificate https://github.com/downloads/linkedin/datafu/datafu-0.0.4.tar.gz $ tar zxvf datafu-0.0.4.tar.gz $ ls datafu-0.0.4/dist datafu-0.0.4-javadoc.jar datafu-0.0.4.jar datafu-docs.war datafu-0.0.4-sources.jar datafu-coverage.war
データの用意
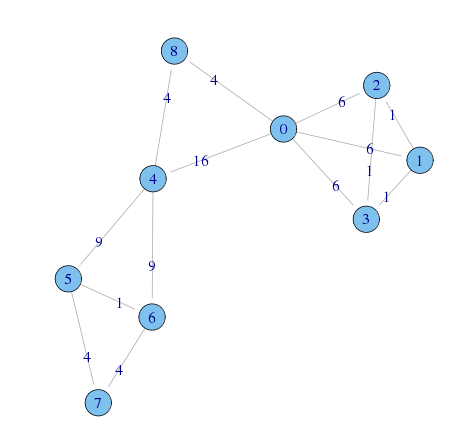
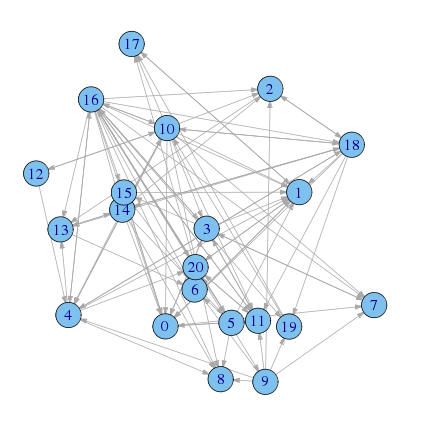
[R]ネットワーク分析 - ネットワークの比較 - yokkunsの日記で使った、ハイテク企業の管理職21人の社会ネットワークの友人ネットワークを使う
library(sna) ## 隣接行列 FRIEND <- matrix(c( 0,1,0,1,0,0,0,1,0,0,0,1,0,0,0,1,0,0,0,0,0, 1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,1, 0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,1,0,0, 1,1,0,0,0,0,0,1,0,0,0,1,0,0,0,1,1,0,0,0,0, 0,1,0,0,0,0,0,0,1,0,1,0,0,1,0,0,1,0,1,0,1, 0,1,0,0,0,0,1,0,1,0,0,1,0,0,0,0,1,0,0,0,1, 0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0, 0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0, 0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0, 0,0,1,0,1,0,0,1,1,0,0,1,0,0,0,1,0,0,0,1,0, 1,1,1,1,1,0,0,1,1,0,0,1,1,0,1,0,1,1,1,0,0, 1,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,1, 0,0,0,0,1,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0, 0,0,0,0,0,0,1,0,0,0,0,0,0,0,1,0,0,0,0,0,0, 1,0,1,0,1,1,0,0,1,0,1,0,0,1,0,0,0,0,1,0,0, 1,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0, 1,1,1,1,1,1,1,1,1,1,1,1,0,1,1,1,0,0,1,1,1, 0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0, 1,1,1,0,1,0,0,0,0,0,1,1,0,1,1,0,0,0,0,1,0, 0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,1,0,0,0, 0,1,0,0,0,0,0,0,0,0,0,1,0,0,0,0,1,1,0,0,0), nrow = 21, byrow = TRUE) ## エッジリストに変換 friend.edge.list <- as.edgelist.sna(FRIEND) ## 出力 write.table(friend.edge.list,file="friend.edgelist.tsv", row.names = FALSE, col.names = FALSE, sep="\t")
HDFSにput
$ hadoop dfs -put friend.edgelist.tsv ./tmp/friend_edgelist
pig実行
pigコード
register 'datafu-0.0.4.jar';
%default friend_edges_dir 'tmp/friend_edgelist'
%default output_dir './tmp/friend_pagerank'
define PageRank datafu.pig.linkanalysis.PageRank('dangling_nodes','true');
friend_edges = load '$friend_edges_dir' as (
source:int,
dest:int,
weight:double);
friend_edges_grouped = foreach (group friend_edges by source) generate
group as source,
friend_edges.(dest,weight) as edges;
friend_ranks = foreach (group friend_edges_grouped all) generate
flatten(PageRank(friend_edges_grouped.(source,edges))) as (source,rank);
friend_ranks = order friend_ranks by source parallel 1;
store friend_ranks into '$output_dir';
結果
1 0.10949851 2 0.15463765 3 0.021636453 4 0.08672039 5 0.026471648 6 0.015206676 7 0.027113214 8 0.046544723 9 0.02419316 10 0.012026792 11 0.032995425 12 0.06756917 13 0.011376936 14 0.03042857 15 0.029928315 16 0.04438733 17 0.05944765 18 0.07817628 19 0.029773977 20 0.016299177 21 0.07556794
異常検知(変化点検出)のパッケージを作ってみた
時系列的な振る舞いの変化点を検出するためのパッケージを作ってみました。
- CRAN: http://cran.r-project.org/web/packages/ChangeAnomalyDetection/
- github: https://github.com/yokkuns/r-AnomalyDetection
Usage
changeAnomalyDetection(x, term = 30, smooth.n = 7, order = c(1, 0, 0), ...)
| x | 時系列の数値ベクトル | |
| term | 学習期間 | |
| smooth.n | 移動平均の期間 | |
| order | arima関数に渡すorder | |
| ... | arima関数に渡すその他パラメータ |
実行例
パッケージ読み込み
library(ChangeAnomalyDetection) library(RFinanceYJ) library(ggplot2) library(reshape2)
試すデータの用意
今回は、RFinanceYJで適当な株価情報を取得して試してみる
- 株価データの取得
stock <- quoteStockXtsData("2432.t", since = "2011-01-01") head(stock) ## Open High Low Close Volume AdjClose ## 2011-01-04 2914 2978 2906 2969 1379000 2969 ## 2011-01-05 2990 2995 2914 2931 1521100 2931 ## 2011-01-06 2949 2952 2919 2932 1086300 2932 ## 2011-01-07 2922 2969 2912 2917 1216000 2917 ## 2011-01-11 2924 2965 2919 2957 1369100 2957 ## 2011-01-12 2958 3045 2957 3025 2080700 3025 stock <- as.data.frame(stock) stock$date <- as.POSIXct(rownames(stock))
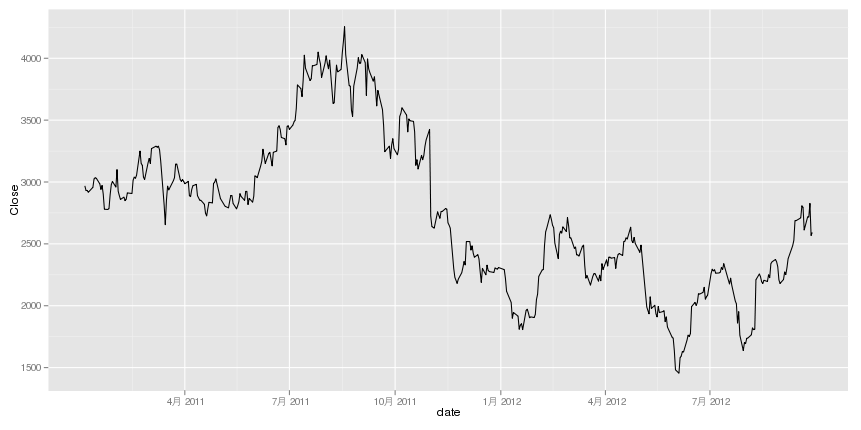
- 終値をプロット
ggplot(yahoo, aes(x = date, y = Close)) + geom_line()
異常スコア算出
- changeAnomalyDetection関数を実行
change.score <- changeAnomalyDetection(x = stock$Close, term = 30, order = c(1, 1, 0)) tail(change.score) ## [1] 0.6212 0.6316 0.6207 0.6011 0.9440 1.1845 stock$change.score <- change.score
- スコアの推移を描画
ggplot(stock, aes(x = date, y = change.score)) + geom_line()
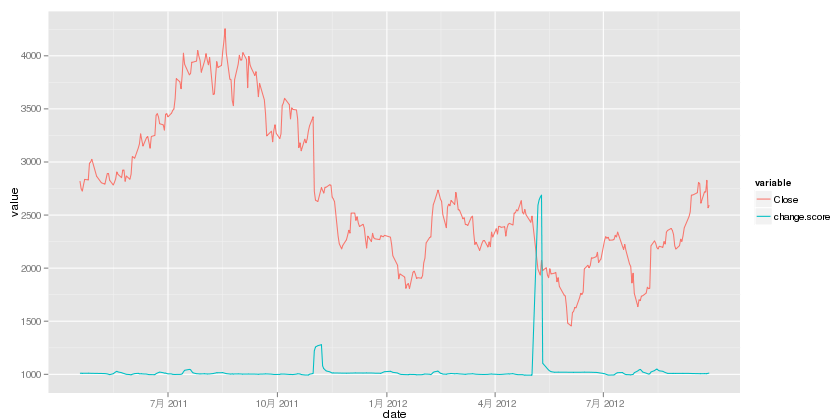
重ねて表示
それぞれで出すと対応が良く分からないので、二つ同時にプロットしてみる。
- データの整形
stock <- stock[stock$change.score != 0, ] # ggplotでは2軸のプロットが出来ないので、スケールを合わせてる stock$change.score <- stock$change.score * 10 + 1000 # この手の整形はreshape2パッケージのmelt関数が便利! stock.melt <- melt(stock, id.vars = "date", measure.vars = c("Close", "change.score"))
- プロット
ggplot(stock.melt, aes(x = date, y = value)) + geom_line(aes(col = variable))
Uplift Modelのパッケージを作ってみた
費用対効果の最大化するためのデータマイニング手法であるUplift ModelをRで実装してパッケージ化してみました。
- Uplift Modelling: 費用対効果の最大化を目的とした最新のデータマイニング手法 - yokkunsの日記
- Google Code Archive - Long-term storage for Google Code Project Hosting.
ドキュメント書くのが面倒で、まだcranにはアップしてませんが、使い方を簡単に紹介したいと思います。
主に使う関数は、以下の2つ
- buildUpliftTree(y.train, treat.train, x.train)
- 学習データから、費用対効果を最大化するような分類を行うモデルを構築する
- classify(uplift.tree, x.new)
- 構築されたUpliftTreeを元に、新しいデータを分類する
ライブラリ読み込み
library(EasyUpliftTree)
データの用意
手元にいい感じのデータがないので、今回はsurvivalパッケージのcolonデータをちょっと加工して使用する。
実際は、医療用のデータだけど、以下のように考えて、y、treat、xを作る。
| status | 購買有無。0: 購入あり、1: 購入なし |
| rx | DM有無。Lev+5FU: DMあり、Obs: DMなし |
| その他 | 何か属性など様々な変数。 |
library(survival) data(colon) sample.data <- na.omit(colon[colon$rx != "Lev" & colon$etype == 2, ]) treat <- ifelse(sample.data$rx == "Lev+5FU", 1, 0) y <- ifelse(sample.data$status == 0, 1, 0) x <- sample.data[, c(4:9, 11:14)] x$v1 <- factor(x$sex) x$v2 <- factor(x$obstruct) x$v3 <- factor(x$perfor) x$v4 <- factor(x$adhere) x$v5 <- factor(x$differ) x$v6 <- factor(x$extent) x$v7 <- factor(x$surg) x$v8 <- factor(x$node4)
学習用のデータとテスト用データの作成
index <- 1:nrow(x) train.index <- index[(index%%2 == 0)] test.index <- index[index%%2 != 0] y.train <- y[train.index] x.train <- x[train.index, ] treat.train <- treat[train.index] y.test <- y[test.index] x.test <- x[test.index, ] treat.test <- treat[test.index]
Uplift Treeの構築
結果ベクトル、介入ベクトル、説明変数を、buildUpliftTree関数に突っ込むだけ
uplift.tree <- buildUpliftTree(y.train, treat.train, x.train) print(uplift.tree) ## sex : 1 ? ## R->nodes : 3 ? ## R->perfor : 1 ? ## R->age : 55.4 ? ## R->1 ## L->1 ## L->nodes : 6 ? ## R->age : 48 ? ## R->v5 : 2 ? ## R->1 ## L->0.75 ## L->nodes : 12.8 ? ## R->1 ## L->0 ## L->v8 : 0 ? ## R->v4 : 0 ? ## R->0.777777777777778 ## L->0 ## L->age : 58.8 ? ## R->0 ## L->0 ## L->v8 : 0 ? ## R->extent : 3 ? ## R->age : 55 ? ## R->v4 : 0 ? ## R->differ : 1.1 ? ## R->0.777777777777778 ## L->0.75 ## L->0.5 ## L->v7 : 0 ? ## R->adhere : 0.199999999999999 ? ## R->1 ## L->1 ## L->0 ## L->0.727272727272727 ## L->0 ## L->age : 63 ? ## R->v2 : 0 ? ## R->nodes : 3.3 ? ## R->age : 67.1 ? ## R->age : 72.8 ? ## R->1 ## L->v8 : 0 ? ## R->1 ## L->0 ## L->0.6 ## L->v5 : 1 ? ## R->extent : 2.5 ? ## R->1 ## L->1 ## L->v5 : 2 ? ## R->v3 : 0 ? ## R->0.733333333333333 ## L->0.5 ## L->age : 72 ? ## R->1 ## L->0 ## L->0.333333333333333 ## L->surg : 1 ? ## R->nodes : 2 ? ## R->0.454545454545455 ## L->0.6 ## L->obstruct : 1 ? ## R->1 ## L->v5 : 1 ? ## R->1 ## L->adhere : 1 ? ## R->1 ## L->age : 48.8 ? ## R->0.166666666666667 ## L->0.142857142857143
購入率の確認
toDateFrame関数でデータフレームに変換して平均を取る
uplift.df <- toDataFrame(uplift.tree) mean(uplift.df[uplift.df$node.type == "R" & uplift.df$treat == 1, "y"]) ## [1] 0.7215 mean(uplift.df[uplift.df$node.type == "L" & uplift.df$treat == 1, "y"]) ## [1] 0.4667
- RグループのDMを受け取ったユーザーの購入率は、Lグループよりもかなり高くなっている
テストデータをRグループとLグループに分類
classify関数に、上で構築したuplift.treeと、新しい説明変数を突っ込む。
x.test$node.type <- sapply(1:nrow(x.test), function(i) classify(uplift.tree, x.test[i, ]))
ROI比較
以下のような条件でROIを計算してみる
- 1人当たりのコスト : 100円
- 1個当たりの売上 : 200円
# 全体にDMを送った場合 roi.data.all <- data.frame(type = "All", cost = length(y.test[treat.test == 1]) * 100, amount = sum(y.test) * 200) # RのグループにDM送った場合 ## Rグループのtreat=0は介入があっても結果が変わらない、Lグループのtreat==1は、介入しなかった場合は、購入無しと厳しめな仮定をおいてる roi.data.r <- data.frame(type = "R", cost = length(y.test[x.test$node.type == "R"]) * 100, amount = sum(y.test[!(x.test$node.type == "L" & treat.test == 1)]) * 200) # ROIを算出 roi.data <- rbind(roi.data.all, roi.data.r) roi.data$ROI <- roi.data$amount/roi.data$cost roi.data ## type cost amount ROI ## 1 All 15000 30200 2.013 ## 2 R 5400 15800 2.926
- Rグループに対してだけDMを送るとROIは2.9倍で、単純に全体に送った場合よりも費用対効果が高くなっている
今後
- 「EasyUpliftTree」という名前の通り、まだまだ簡易版って感じなので、これをたたき台としてiAnalysisの@isseing333さんと開発を進めていこうと思います!
第26回TokyoRでData Fusionについて発表しました
ネットワーク構造の分析 - コミュニティの抽出
ある程度の規模のネットワークでは、内部にサブネットワーク(コミュニティ)が形成されることがある
例えば、大学のネットワーク図を描くと、何となく学部だったりサークルのグループが見えてくる
このよなコミュニティの抽出方法として、辺の媒介中心性を用いた方法があるので、その方法とRでの実行を紹介する
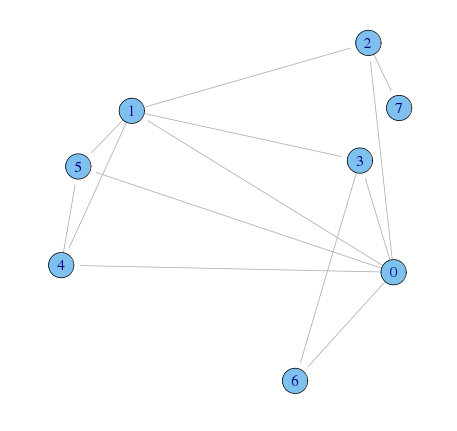
データの入力と描画
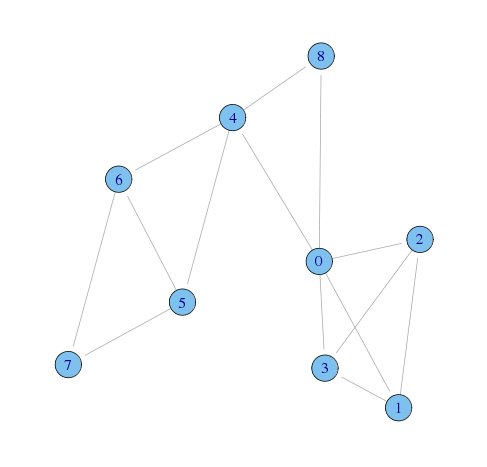
g <- graph(c( 1,2, 1,3, 1,4, 1,5, 1,9, 2,3, 2,4, 3,4, 5,6, 5,7, 5,9, 6,7, 6,8, 7,8) - 1, n = 9, directed = FALSE)
plot(g,layout=layout.lgl)
何となく、以下のようなコミュニティがありそう
辺の媒介中心性
[R][ネットワーク分析] ネットワークにおいてどれくらい中心的かの指標 - yokkunsの日記の媒介中心性を、エッジに適用したもの。
ある人とある人のつながりを除外すると、コミュニティ間のつながりがなくなったり、遠くなるようなつながりのスコアが高くなる
Rでは、igraphパッケージのedge.betweenness関数で計算出来る
(g.edge.betweenness <- edge.betweenness(g, directed=FALSE)) [1] 6 6 6 16 4 1 1 1 9 9 4 1 4 4
エッジに媒介中心性の値を入れると以下のような感じ
plot(g,layout=layout.fruchterman.reingold,edge.label=g.edge.betweenness)
辺の媒介中心性による分割
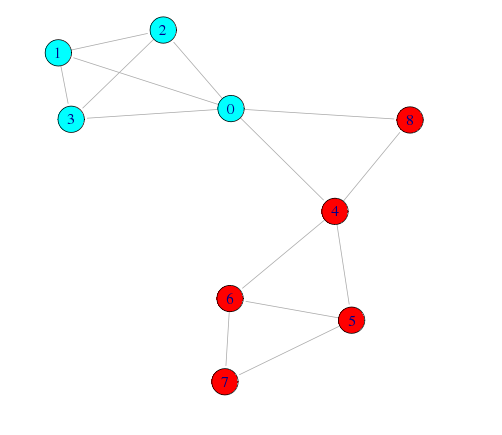
媒介中心性が最大になるエッジを取り除いて分割、というのを繰り返す
> (eb <- edge.betweenness.community(g)) $removed.edges [1] 3 4 10 8 9 0 1 2 5 6 7 11 12 13 $edge.betweenness [1] 16.0 20.0 4.0 1.5 3.0 1.0 1.5 3.0 1.0 2.0 1.0 1.0 2.0 1.0 $merges [,1] [,2] [1,] 6 7 [2,] 5 9 [3,] 2 3 [4,] 1 11 [5,] 0 12 [6,] 4 10 [7,] 14 8 [8,] 13 15 $bridges [1] 14 13 11 10 8 5 3 2 attr(,"class") [1] "igraph.ebc"
eb$mergeは各ノードが合同してコミュニティを形成してく過程を示している。
最初は各ノードが1つのコミュニティの状態からはじまり、6と7がつながって9というコミュニティを生成、次に、5とそのコミュニティが合同して・・・という流れ。
この様子は、デンドログラムの方が分かりやすい
dend <- as.dendrogram(eb) plot(dend)
デンドログラムを任意のステップ数で切れば、いくつかのコミュニティが形成される
見方としては以下のような感じで、各ステップ数でのコミュニティが分かる
モジュラリティQ
辺の媒介中心性を用いることで、任意のコミュニティ抽出が出来る事が分かったが、どのステップ数が最適なのかは分からない
何をもって最適とするかは難しいが、一つの手がかりとして、分割されたコミュニティ内のつながり具合と、コミュニティ間のつながり具合を比較するモジュラリティQという指標がある
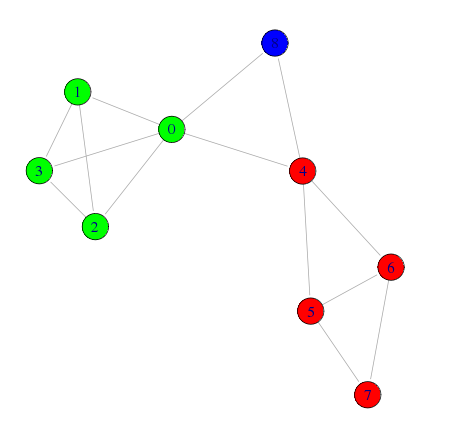
とりあえず、モジュラリティQが最大になる分割
# モジュラリティQが最大になる分割 wt <- walktrap.community(g,modularity=T) memb <- community.to.membership(g, wt$merges, steps=which.max(wt$modularity)-1) # コミュニティ毎に色を設定 V(g)$color <- rainbow(length(memb$csize))[memb$membership+1] # ネットワーク図の描画 plot(g, layout=layout.fruchterman.reingold, edge.arrow.size=0.5)
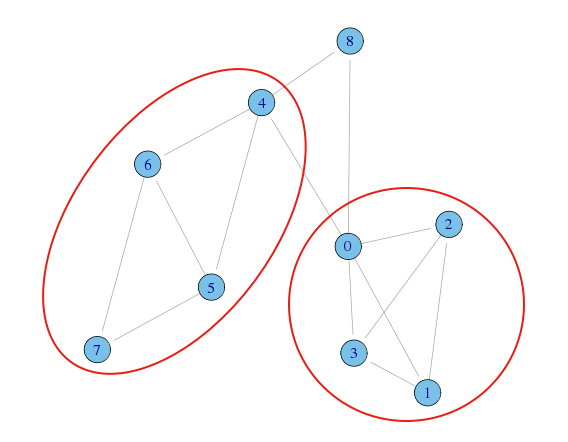
任意の数で分割
モジュラリティQは、一つの手がかりにはなるが、普遍的な妥当性を持っている訳でない
今回の例では、ネットワーク図やデンドログラムを見ると、8番は別のコミュニティ(1人だけど)として見た方が自然だと思われる
# 6ステップでコミュニティ分割 memb2 <- community.to.membership(g, eb$merges, steps=6) # コミュニティ毎に色を設定 V(g)$color <- rainbow(length(memb2$csize))[memb2$membership+1] # ネットワーク図の描画 plot(g, layout=layout.fruchterman.reingold, edge.arrow.size=0.5)
[R][ネットワーク分析] ネットワークにおいてどれくらい中心的かの指標
ネットワーク分析で最も良く用いられる指標として、中心性というものがある
今回は、その中でも3つの指標に絞って紹介
次数中心性
友達が多い人が高く評価される指標
友達の数を数えるだけなので、計算も簡単
- データの入力と描画
A <- matrix(c( 0,1,1,1,1,1,1,0, 1,0,1,1,1,1,0,0, 1,1,0,0,0,0,0,1, 1,1,0,0,0,0,1,0, 1,1,0,0,0,1,0,0, 1,1,0,0,1,0,0,0, 1,0,0,1,0,0,0,0, 0,0,1,0,0,0,0,0), nrow = 8, byrow = TRUE) g <- graph.adjacency(A, mode = "undirected") plot(g)
- 次数中心性の算出
> degree(g) [1] 6 5 3 3 3 3 2 1
PageRank
単純に友達が多いだけではなく、その友達の友達の数も考慮した指標
たくさん友達はいるんだけど、自分以外の友達がいない人とばかりつながっている人と、同じ数の友達がいて、その友達にもまたたくさん友達がいる人とでは、やっぱり後者の方が重要だと考えられる
- データの入力と描画
A <- matrix(c( 0,0,0,0,0,0,0,0,0, 1,0,0,0,0,0,0,0,0, 1,0,0,0,0,0,0,0,0, 0,1,1,0,0,0,0,0,0, 1,0,0,0,0,0,0,0,0, 0,0,0,0,0,0,0,0,0, 0,0,0,0,0,1,0,0,0, 0,0,0,0,0,1,0,0,0, 0,0,0,0,0,1,0,0,0), nrow = 9, byrow = TRUE) g <- graph.adjacency(A, mode = "directed") plot(g,edge.arrow.size=.1)
- Page Rankの算出
> page.rank(g, directed = TRUE)$vector [1] 0.40153809 0.07480774 0.07480774 0.03799515 0.03799515 0.25887070 0.03799515 0.03799515 [9] 0.03799515
-
- 0番と5番の人は、同じ次数だが0番の人の方がスコアが高くなっている
媒介中心性
その人がいないと情報が伝わらなかったり、おそくなったりするような位置の人を重要とする指標
複数のコミュニティに所属してるような人が該当する (その人がいないと、コミュニティ間のつながりが薄くなるような人)
- データの入力と描画
A <- matrix(c( 0,1,1,1,1,1,1,0, 1,0,1,1,1,1,0,0, 1,1,0,0,0,0,0,1, 1,1,0,0,0,0,1,0, 1,1,0,0,0,1,0,0, 1,1,0,0,1,0,0,0, 1,0,0,1,0,0,0,0, 0,0,1,0,0,0,0,0), nrow = 8, byrow = TRUE) g <- graph.adjacency(A, mode = "undirected") plot(g)
- 媒介中心性の算出
> betweenness(g) [1] 8.5 4.0 6.0 0.5 0.0 0.0 0.0 0.0
参考資料
[R][ネットワーク分析] ネットワーク構造の諸指標
ネットワーク分析 (Rで学ぶデータサイエンス 8)の第3章の内容
密度(density)
グラフにおいて張ることの出来る全てのエッジの数に対する、実際のエッジの数の比率
例
- データの入力
library(igraph) A <- matrix(c( 0,1,1,1,1, 1,0,0,1,1, 1,0,0,0,0, 1,1,0,0,0, 1,1,0,0,0),nrow=5) B <- matrix(c( 0,1,1,1,1, 0,0,0,1,1, 0,0,0,0,0, 0,0,0,0,0, 0,0,0,0,0),nrow=5,byrow=T) g1 <- graph.adjacency(A, mode = "undirected") g2 <- graph.adjacency(B)
- Aのプロット
plot(g1)
- Bのプロット
plot(g2)
- 密度の計算
> graph.density(g1) [1] 0.6 > graph.density(g2) [1] 0.3
推移性(transitivity)
ネットワークにおいて、ノードiとノードjの間、及びノードjとノードkの間に関係が合って、ノードiとノードkの間も関係がある場合、関係が推移的(transitive)であるという
例えば、「自分の友達の友達が、自分の友達でもある」というように、関係が三角形を作っている場合がこれにあたる
推移性(transitivity)とは、推移的な関係が成り立っている程度を比率で表したもの
- データの入力
A <- matrix(c( 0,1,1,1,1,1,0,0, 1,0,1,0,0,0,1,0, 1,1,0,0,0,0,0,0, 1,0,0,0,1,0,0,1, 1,0,0,1,0,1,0,0, 1,0,0,0,1,0,0,0, 0,1,0,0,0,0,0,0, 0,0,0,1,0,0,0,0),nrow=8) g <- graph.adjacency(A, mode="undirected")
- Aのプロット
plot(g)
- 推移性の計算
> transitivity(g) [1] 0.4285714
相互性(reciprocity)
有向グラフ全体において、「両想い」がどらくらいの割合を占めているか
- データの入力
A <- matrix(c( 0,1,0,0,1, 1,0,1,0,0, 0,1,0,1,0, 1,0,1,0,0, 0,0,0,1,0),nrow=5,byrow=T) g <- graph.adjacency(A)
- Aのプロット
plot(g)
- 相互性の計算
> reciprocity(g) [1] 0.5
分析例
データは、第8章のハイテク企業の管理職21人の友人ネットワーク
データの入力
> (friend <- read.csv("friend.csv",header=F)) V1 V2 V3 V4 V5 V6 V7 V8 V9 V10 V11 V12 V13 V14 V15 V16 V17 V18 V19 V20 V21 1 0 1 0 1 0 0 0 1 0 0 0 1 0 0 0 1 0 0 0 0 0 2 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 1 3 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 1 0 0 4 1 1 0 0 0 0 0 1 0 0 0 1 0 0 0 1 1 0 0 0 0 5 0 1 0 0 0 0 0 0 1 0 1 0 0 1 0 0 1 0 1 0 1 6 0 1 0 0 0 0 1 0 1 0 0 1 0 0 0 0 1 0 0 0 1 7 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 8 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 9 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 10 0 0 1 0 1 0 0 1 1 0 0 1 0 0 0 1 0 0 0 1 0 11 1 1 1 1 1 0 0 1 1 0 0 1 1 0 1 0 1 1 1 0 0 12 1 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 1 13 0 0 0 0 1 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 14 0 0 0 0 0 0 1 0 0 0 0 0 0 0 1 0 0 0 0 0 0 15 1 0 1 0 1 1 0 0 1 0 1 0 0 1 0 0 0 0 1 0 0 16 1 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 17 1 1 1 1 1 1 1 1 1 1 1 1 0 1 1 1 0 0 1 1 1 18 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 19 1 1 1 0 1 0 0 0 0 0 1 1 0 1 1 0 0 0 0 1 0 20 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 1 0 0 0 21 0 1 0 0 0 0 0 0 0 0 0 1 0 0 0 0 1 1 0 0 0 > g <- graph.adjacency(friend)
推移性
> transitivity(g) [1] 0.4714946
- 「自分の友達の友達が、自分の友達(片想い含む)」という関係が半分くらい
相互性
> reciprocity(g) [1] 0.2911392
- 互いに友達と思っている関係は、3割程度しかないという悲しい現実